Современная биотехнология неразрывно связана с использованием новых подходов к отбору природных микроорганизмов при создании бактериальных препаратов.
Фундаментальные достижения и практические исследования последних десятилетий в микробиологии, генетике и молекулярной биологии дали возможность изучения генетического разнообразия микроорганизмов. С развитием генетической систематики, разнообразием используемых методов, направленных на изучение бактериального генома и накоплением экспериментальных данных, позволило решить многие спорные вопросы систематики конкретных групп микроорганизмов [10].
До недавнего времени при отборе штаммов для создания бактериальных консервантов использовались в основном классические микробиологические методы. Оценки стартерных культур, такие как выделение, идентификация таксономического положения, проводились на основе изучения их морфологических, культуральных, физиолого-биохимических и технологических свойств. Однако применение только традиционных способов не всегда позволяет эффективно отбирать безопасные и хозяйственно-ценные штаммы молочнокислых бактерий для использования их в качестве биологического агента при ферментации растительной массы.
В связи с этим целью данной работы явилось выделение чистых культур молочнокислых бактерий из различных природных субстратов и определение видовой принадлежности полученных лактобацил классическимии молекулярно-генетическими методами.
Материалы и методы исследования
Источники выделения. В качестве источников выделения изолятов молочнокислых бактерий использовали филосферу многолетних бобовых трав (клевер луговой – TrifoliumpratenseL., сорт Ранний-2 и люцерна изменчивая – MedicagovariaMartyn, сорт Айслу) и растительный сок из надземной части указанных культур.
Навески проб (5г) надземной части (филосфера) вегетирующих бобовых растений помещали в 45 мл стерильной воды и интенсивно встряхивали на качалке при 120 об/мин в течение 30 мин. Для получения растительного сока бобовых растений использовали листья с черешками и фрагментами стеблей, измельченные до 2–3 см, и растительную массу отжимали с помощью пресса. Полученную субстанцию несколько раз фильтровали через стерильную ткань.
Культивирование микроорганизмов. Из полученных биологических материалов готовили серию разведений и высевали на агаризованные среды. Среду сусло-агар (5° по Баллингу) с 2 % карбонатом кальция использовали для выделения молочнокислых изолятов и отбирали колонии с различной зоной растворения мела вокруг них, которая косвенно служила признаком активности кислотообразования изолята молочнокислой бактерии [1,8]. Далее бактериальные изоляты культивировали на жидкой питательной среде MRS[15] с последующим поверхностным пересевом их на агаризованную среду MRS. Среди морфологически идентичных колоний (по окраске, форме, текстуре) для выделения в чистую культуру выбирали одну и пересевали на плотную селективную среду Рогозы [18] с последующей идентификацией молочнокислых бактерий. Культивировали молочнокислые бактерии при 37 °С.
Классическая идентификация. Идентификацию молочнокислых бактерий проводили на основе физиолого-биохимических тестов [13,6,11]. Тесты на каталазную активность, восстановление нитратов, сбраживание сахаров, газообразование, рост при различных температурах и другие признаки определяли с применением стандартных методов [5,3].
Молекулярно-генетическая идентификация. Для проведения молекулярно-генетической идентификации отбирали одиночные колонии исследуемых штаммов молочнокислых бактерий, выросшие на плотной питательной среде Рогозы в течение 24 часов при температуре 37 °С. Бактериальные клетки переносили в 1 мл стерильного физиологического раствора, ресуспендировали и осаждали их центрифугированием при 13 000 об/мин в течение 2 мин. Выделение бактериальной ДНК осуществляли по методу, описанному в работе [2], увеличив рабочую концентрацию лизоцима (Serva, Германия) в лизирующем буфере до 1мг/мл. Амплификацию фрагментов генов 16SрРНК и pheS проводили с помощью полимеразной цепной реакции (ПЦР) в смеси финальным объемом 50 мкл, включавшей 5 мкл 20 мМ раствора dNTP, 5 мкл 10x ПЦР буфера, 0.4 мкл 5 ед. акт. Taq ДНК-полимеразы, 1 мкл 50 мкМ каждого смыслового и антисмыслового праймеров, 1 мкл препарата бактериальной ДНК. Для амплификации фрагмента гена 16SрРНК использовали универсальные прокариотические праймеры 16S-8F 5’-agagtttgatcctggctcag и 16S-1492R5’-ggttaccttgttacgactt [20]. Для секвенирования использовали праймеры 16S-529R 5’-acgcttgccacctacgtattac, 16S-806F 5’-ggactaccagggtatctaat и 16S-U1Rm 5’-gggcccccgtcaattcctttgag. При синтезе фрагмента гена pheS и при осуществлении секвенирования применяли вырожденные олигонуклеотидыpheS-21-F 5’-cayccngchcgygayatgc и pheS-22-R5’-ccwarvccraargcaaarcc [16]. Амплификацию проводили на приборе «MastercyclerGradient» (Eppendorf, Германия) по протоколам, описанным [20,16]. Секвенирование ПЦР-продуктов проводили в лаборатории ЗАО «ЕврогенРу» (Россия) на генетическом анализаторе «ABIPRISM 3100» (AppliedBiosystems, США). Результаты обрабатывали с помощью программы BioEdit 7.1.3.0. Процедуру множественного выравнивания последовательностей проводили по алгоритму CrustalW. Идентификацию видовой принадлежности осуществляли путем сравнения полученных последовательностей с базой данных GenBank (http://www.ncbi.nlm.nih.gov/blast) (NCBI, США). Расчет величин эволюционных дистанций и филогенетический анализ проводили с помощью программы Mega 5.1. согласно рекомендациям [4].
Идентификация микроорганизмов методом MALDI–TOFMS. Определение видовой принадлежности изучаемых микроорганизмов проводили на приборе MicroflexMaldiBioTyper (BrukerDaltonics, Германия). Изолированные колонии микроорганизмов, выросшие на поверхности агаризованной питательной среды Рогозы. Анализ масс-пик листов рибосомальных белковых спектров осуществляли с помощью программного обеспечения MALDI Biotyper 3.1 (BrukerDaltonics, Германия). В качестве сравниваемых штаммов использовали источники из коллекции микроорганизмов DMZS (Германия), выбранные фирмой-производителем при создании прибора. Интерпретацию результатов проводили по шкале, предлагаемой фирмой-производителем: показатель Score с величиной равной 2.0 и более считали надежным для определения вида, в диапазоне от 1.7 до 2.0 надежным для определения рода, показатель менее 1.7 свидетельствовал о ненадежной идентификации.
Продукты брожения анализировали методом высокоэффективной жидкостной хроматографии (ВЭЖХ) на приборе Series-200 (PerkinElmer», США) с колонкой С18 (250×4.6 мм), используя рефрактометрический детектор. Пробы готовили с помощью картриджей SerPakC-18 (TESSEK, Чехия), заполненных сорбентом Sеpаrоn марки SGX-С18 (зернение 60мкм, диаметр пор 80А). Элюцию проводили 0.1 % раствором ортофосфорной кислоты при температуре 30 ºС и скорости протока элюента 0.6 мл/мин. В качестве стандартов использовали водные растворы органических кислот («ACROS», Бельгия).
В работе использовались стандартные методы математической статистики с применением пакета прикладных программ MS Excel.
Результаты исследования и их обсуждение
Применение современных молекулярно-биологических и генотипических подходов при таксономической идентификации молочнокислых бактерий позволяет выбирать стартовые культуры с прогнозируемыми признаками, необходимые для разработки биологических консервантов.
Впервые в условиях Республики Татарстан выделены изоляты молочнокислых бактерий из природных источников, надземной части бобовых культур (филосфера, растительный сокклевера и люцерны). Начальным этапом наших исследований была индикация и идентификация изолятов молочнокислых бактерий классическими микробиологическими методами. С использованием селективной среды Рогозы осуществили тестирование более 40 изолятов бактерий, что позволило получить 15 изолятов с характерными свойствами возбудителей молочнокислого брожения. Из них, с учетом типа брожения (предпочтение отдано гомоферментативному), скорости и масштабов ацидогенеза, проведен первичный отбор семи изолятов. Темпы и уровень кислотообразования указанных изолятов были подтверждены ВЭЖХ анализом органических кислот. В итоге, по совокупности результатов дифференциально-диагностического тестирования [13,14,11] изоляты отнесены к семейству Lactobacillaceae, роду Lactobacillus. Для дальнейших исследований были отобраны 4 штамма молочнокислых бактерий. Результаты фенотипической идентификации полученных четырех штаммов представлены в таблице 1.
|
Свойства |
Штаммы молочнокислых бактерий |
|||
|
RS3 |
RS4 |
RS6 |
RS7 |
|
|
сбраживание углеводов: |
||||
|
глюкоза |
+ |
+ |
+ |
+ |
|
фруктоза |
+ |
+ |
+ |
+ |
|
мальтоза |
+ |
+ |
+ |
+ |
|
сахароза |
+ |
+ |
+ |
+ |
|
галактоза |
+ |
+ |
+ |
+ |
|
арабиноза |
– |
– |
– |
– |
|
рамноза |
– |
– |
– |
– |
|
ксилоза |
– |
– |
– |
– |
|
сорбит |
– |
– |
– |
– |
|
биохимические: |
||||
|
рост при 15ºС |
– |
– |
– |
– |
|
рост при 45ºС |
– |
– |
– |
– |
|
каталаза |
– |
– |
– |
– |
|
восстановление NOˉ3 |
– |
– |
– |
– |
|
газообразование из глюкозы |
– |
– |
– |
– |
|
лактат, мг/мл |
18.3 |
14.4 |
11.1 |
12.9 |
|
ацетат, мг/мл |
3.0 |
1.6 |
0.9 |
1.3 |
Таблица 1
Классические идентификационные признаки природных штаммовмолочнокислых бактерий
|
Свойства |
Штаммы молочнокислых бактерий |
|||
|
RS3 |
RS4 |
RS6 |
RS7 |
|
|
морфологические: |
||||
|
форма клеток |
палочки |
палочки |
палочки |
палочки |
|
окраска по Граму |
+ |
+ |
+ |
+ |
|
кислотоустойчивость |
+ |
+ |
+ |
+ |
|
спорообразование |
– |
– |
– |
– |
|
подвижность |
– |
– |
– |
– |
|
культуральные: |
||||
|
форма колоний |
мелкие выпуклые |
мелкие выпуклые |
мелкие выпуклые |
мелкие выпуклые |
|
цвет колоний |
белые матовые |
белые матовые |
бесцветные |
бесцветные |
|
рост на жидких средах |
придонный |
придонный |
равномерный рост по всей толще среды |
придонный |
+ , активный рост; –, отсутствие роста.
Материалы,отображающие источники происхождения штаммов, представлены в таблице 2.
Таблица 2
Происхождения природных штаммов молочнокислых бактерий
|
№ |
Наименованиештамма |
Источник выделения |
|
1. |
Lactobacillussp.RS3 |
филосфера люцерны изменчивой(MedicagovariaMartyn) |
|
2. |
Lactobacillussp. RS4 |
филосфера клевера лугового (TrifoliumpratenseL.) |
|
3. |
Lactobacillussp.RS6 |
растительный сок клевера лугового (TrifoliumpratenseL.) |
|
4. |
Lactobacillussp.RS7 |
растител. сок люцерны изменчивой (MedicagovariaMartyn) |
Штаммы молочнокислых бактерий депонированы во Всероссийской коллекции микроорганизмов (ВКМ) Института биохимии и физиологии микроорганизмов им. Г.К. Скрябина под номерами: ВКМВ-2342 ДLactobacillussp. RS3,ВКМВ-2343 ДLactobacillus sp. RS4, ВКМВ-2344 ДLactobacillussp. RS7.
Следует отметить, что применение указанных методов, предусматривающих выделение чистой культуры и определение морфологических, культуральных признаков, спектра сбраживаемых углеводов и биохимических свойств изолятов молочнокислых бактерий, было затратным и требовало достаточно длительного времени для завершения работы и получения результатов. Однако исследования показали, что идентификация молочнокислых бактерий только на основании вышеперечисленных признаков в настоящее время является недостаточной. Так, согласно работам авторов [12,9], многие виды молочнокислых бактерий обладают высоким уровнем фенотипической изменчивости под воздействием различных факторов.
В связи с этим мы продолжили типирование наших природных штаммов молочнокислых бактерий современными молекулярно-биологическими методами для таксономической идентификации. В результате секвенирования штаммов молочнокислых бактерий определены нуклеотидные последовательности гена 16SрРНК длиной 1437 п.н. Полученные нуклеотидные последовательности были использованы при проведении филогенетического анализа. В качестве сравнения использовали нуклеотидные последовательности генов 16SрРНКреферентных штаммов основных видов рода Lactobacillus, полученных из базы данных GenBank (NCBI, США).
Проведенный анализ показал, что идентифицируемые четыре штамма природных молочнокислых бактерий располагались в одном кластере с референтными штаммами, относящимися к видам Lactobacillusplantarum, L. paraplantarum и L. pentosus (рис. 1). На основании этого было сделано заключение, что типируемые штаммы относятся к группе видов близких к Lactobacillusplantarum.

Филогенетическое положение природных штаммов лактобацилл на основании анализа нуклеотидных последовательностей фрагмента гена 16SрРНК. Расчет величин эволюционных дистанций проведен по модели Джукса – Кантора. Построение дендрограммы осуществлено согласно алгоритму минимума эволюции.
Знаком «♦» – отмечены идентифицируемые штаммы.
Согласно литературным данным различие в нуклеотидных последовательностях генов 16SрРНК данных видов составляет менее 1 % [17], что не позволяет проводить окончательную надежную видовую идентификацию по данному молекулярному признаку.
Был проведен молекулярно-генетический анализ нуклеотидной последовательности фрагмента гена pheS размером 454 п.н. В ходе филогенетического анализа была выявлена локализация всех четырех идентифицируемых в одном кластере с референтными штаммами, относящимися к видам Lactobacillusplantarum (рис.2). Примененный подход позволил достоверно дискриминировать идентифицируемые штаммы от видов Lactobacillusparaplantarum и L. pentosus. В связи с этим штаммы RS3, RS4, RS6 и RS7 были отнесены к виду Lactobacillusplantarum.
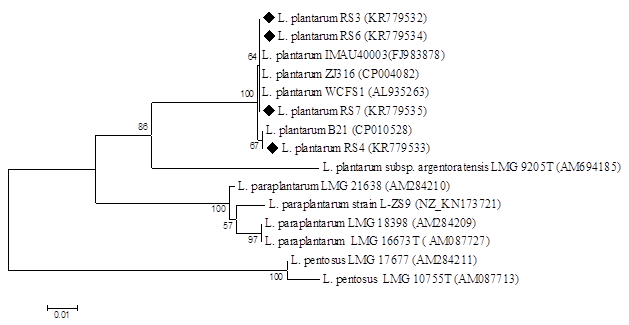
Филогенетическое положение природных штаммов лактобацилл на основании анализа нуклеотидных последовательностей фрагмента гена pheS. Расчет величин эволюционных дистанций проведен по модели Джукса – Кантора. Построение дендрограммы осуществлено согласно алгоритму минимума эволюции.
Знаком «♦» – отмечены идентифицируемые штаммы.
Нуклеотидные последовательности данных штаммов аннотированы в базе данных GenBankNCBI под следующими номерами: L. plantarumRS3 – KR779536 и 779532, L. plantarumRS4 – KR779537 и KR779533, L. plantarumRS6 – KR779538 и KR779534, L. plantarumRS7 – KR 779539 и KR779535.
С разработкой метода MALDI-TOFMS и накоплением мирового опыта его применения для видовой идентификации микроорганизмов стало возможным быстро и с высокой пропускной способностью типировать их из микробной популяции в биологических субстратах практически без предварительной подготовки образца [7,19].
Применение метода MALDI–TOF MS подтвердило результаты проведенной молекулярно-генетической идентификации видовой принадлежности молочнокислых бактерий. Максимальные величины показателя Score были отмечены при сравнении с референтными штаммами, относящимися к виду Lactobacillusplantarum (табл. 3).
Таблица 3
Результаты масс-спектрометрической идентификации природных штаммов молочнокислых бактерий
|
№
|
Референтные штаммы |
ПоказательScore идентифицируемых штаммов |
|||
|
L. RS3 |
L. RS4 |
L. RS6 |
L. RS7 |
||
|
1. |
Lactobacillus plantarum DSM 1055 |
2.19 |
2.32 |
2.28 |
2.23 |
|
2. |
L. plantarumDSM 20246 |
2.25 |
2.31 |
2.27 |
2.18 |
|
3. |
L. plantarumDSM 2601 |
2.15 |
2.29 |
2.28 |
2.17 |
|
4. |
L. plantarumssp. argentoratensis DSM 16365 |
1.87 |
2.01 |
2.03 |
1.89 |
|
5. |
L. pentosusDSM 20314 |
1.93 |
1.94 |
1.90 |
1.85 |
|
6. |
L. paraplantarumDSM 10667 |
1.66 |
1.98 |
1.76 |
1.75 |
Проведенное исследование обосновало достоверность и воспроизводимость результатов идентификации микроорганизмов с помощью время-пролётной масс-спектрометрии и показало целесообразность ее внедрения в практику типирования для видовой идентификации ряда грамположительных молочнокислых бактерий. Результаты исследований показывают, что группа бактерий Lactobacillusplantarum, основанные на профилях MALDI-TOFMS, коррелируют с филогенетическими генными группами pheSи16SrRNA. При этом метод MALDI–TOFMS оказался наиболее эффективным и достоверным, быстрым и экономичным способом видовой идентификации природных молочнокислых бактерий.
Заключение
Таким образом, таксономическую идентификацию чистых культур молочнокислых бактерий, выделенных из различных природных субстратов (надземная часть бобовых культур), целесообразно проводить с привлечением микробиологических и молекулярно-генетических методов. Такой комплексный подход позволяет идентифицировать штаммы по совокупности описанных признаков на видовом уровне, что в дальнейшем обеспечивает создание на их основе эффективных биологических консервантов с прогнозируемыми биотехнологическими свойствами.
Рецензенты:
Хайруллин Р.М., д.б.н., профессор, главный научный сотрудник ФГБУН Института биохимии и генетики Уфимского научного центра РАН, г. Уфа;
Дегтярева И.А., д.б.н., заведующий отделом агроэкологии и микробиологии ФГБНУ «Татарский научно-исследовательский институт агрохимии и почвоведения», г. Казань.



