E. coli M-17 имеет долгую историю использования в качестве продуцента пробиотического препарата «Колибактерин сухой», на протяжении которой штамм неоднократно подвергался многочисленным исследованиям с целью определения способности к продукции колицинов и уровня антагонистической активности, антибиотикорезистентности, биохимической активности и т.д. Безвредность штамма была подтверждена эмпирическим путем. В начале использования данного штамма в качестве продуцента пробиотика важное диагностическое значение имел его серотип (О2:Н6), так как штамм периодически проводили через организм здоровых добровольцев для сохранения его антагонистических свойств [4].
Согласно требованиям современных нормативных документов производственные пробиотические штаммы должны быть изучены по множеству признаков, в том числе подтверждающих их безвредность, апатогенность и авирулентность [1; 2]. Существующие в настоящее время наукоемкие технологии позволяют провести анализ полного генома штаммов микроорганизмов и подтвердить их безвредность на генетическом уровне.
Целью нашей работы было углубленное исследование биологических свойств штамма E. coli М-17, использующегося для производства пробиотика «Колибактерин сухой» с использованием наукоемких методов – современных биохимических тест-систем, MALDI TOF масс-спектрометрии, полногеномного секвенирования и биоинформатического анализа данных.
Материалы и методы исследования. Восстановление лиофильно высушенного штамма проводили с использованием мясо-пептонного бульона (Nutrient Broth, HiMedia), подращённую культуру титровали в диапазоне разведений 10-1 – 10-7 и проводили высевы по 0,05 мл на среду Эндо (Питательная среда для выделения энтеробактерий – агар Эндо ГРМ, Оболенск), далее инкубировали при 37 °С 24 часа. Выросшие колонии микроорганизмов оценивали по морфологии, по 10 колоний каждого морфологического вида отбирали для следующего этапа исследования – масс-спектрометрии.
Масс-спектрометрический анализ осуществляли с помощью времяпролетного MALDI масс-спектрометра Autoflex (Bruker Daltonics, Германия), пробоподготовка суточных культур исследуемых микроорганизмов проводилась методом прямого нанесения по стандартному протоколу, представленному в руководстве пользователя. Идентификация, запись, обработка и анализ масс-спектров проводилась с помощью программы BioTyper RTC.
Остаток колонии засевали на мясо-пептонный бульон для наращивания биомассы и последующей биохимической идентификации. Для биохимической идентификации отбирали культуры, по результатам масс-спектрометрии имевшие значения Score 2,100 и более.
Для расширенного изучения биохимических свойств штаммов использовали пластины биохимические, дифференцирующие энтеробактерии (ПБДЭ) («Диагностические системы», Россия), системы индикаторные бумажные для идентификации микроорганизмов - набор № 2 для межродовой и видовой дифференциации энтеробактерий (ФГУП НПО «Микроген» Минздрава России) и среды HiMedia (HiMedia Laboratories Pvt. Limited (Индия)). Пробоподготовку, культивирование, идентификацию микроорганизмов и интерпретацию полученных результатов проводили согласно инструкциям производителей.
В работу по полногеномному секвенированию была отобрана культура E. coli M-17 с изученным профилем рибосомальных белков с максимально высоким Score, известной биохимической активностью. Геномную ДНК выделяли с использованием коммерческого набора QIAamp DNA Mini Kit (QIAGEN, Германия), подготовку библиотек производили с помощью набора TrueSeq (Illumina Inc, США), секвенирование выполняли на платформе MiSeq (Illumina). Аннотация генома производилась с помощью утилиты Prokka v. 1.11 [17] и геномного сервера RAST [16]. Для подробного изучения генома использовали специализированные программные продукты, доступные на сайте Центра геномной эпидемиологии: изучение CRISPR-региона проводили с использованием программы CrisprFinder [10], поиск детерминант антибиотикорезистентности и патогенности с использованием ResFinder 2.1 и PathogenFinder [9; 18]. Для поиска детерминант вирулентности применяли программу VirulenceFinder [11], для обнаружения интегрированных плазмид – PlasmidFinder 1.3. [8]. MLST типирование проводили с использованием программы MLST-1.8 Server [13], для установления серотипа использовали программу SerotypeFinder 1.1 [12].
Результаты исследования и их обсуждение. Для идентификации методом масс-спектрометрии были выбраны характерные крупные малиновые колонии с металлическим блеском в S-форме. В результате идентификации микроорганизмов в автоматическом режиме получен ряд масс-спектров со Score от 2,05 до 2,56. Характерный масс-спектр представлен на рис. 1.
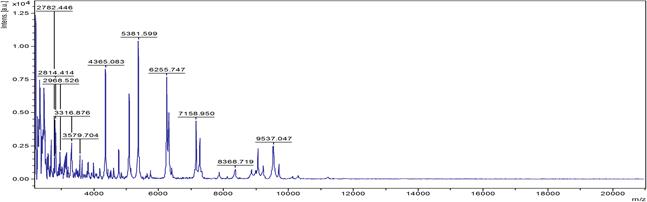
Рис. 1. MALDI масс-спектр штамма E. coli M-17 при использовании α-CHCA матрицы
Установлено, что масс-спектр штамма E. coli М-17 составляют 85 пиков, 62 из которых воспроизводимы. При анализе ряда полученных масс-спектров обнаружено, что для изучаемого штамма характерно наличие следующих масс ионизированных белков (m/z): 2329, 2569, 2940, 3168, 3638, 5340, 6243, 6255, 6276, 8875, 9713.
В результате исследования биохимических с помощью стандартных тест-систем ПБДЭ и СИБ, а также дополнительных тестов (ксилоза, рамноза, дульцит и тест на желатиназную активность) с использованием сред HiMedia выявлено, что данный штамм обладает способностью ферментировать сахарозу, мальтозу, сорбит, глюкозу, лактозу, арабинозу, маннит, ксилозу, рамнозу, образует индол, обладает β-галактозидазной активностью, утилизирует цитрат натрия с глюкозой, образует лизин- и орнитиндекарбоксилазу. Штамм неспособен разжижать желатин, утилизировать цитрат и малонат натрия, не обладает аргининдегидролазой, фенилаланиндезаминазой, не образует ацетилметилкарбинол, не ферментирует инозит, дульцит, не гидролизует мочевину, не образует сероводород. Именно такой биохимический профиль характерен для классического штамма E. coli M-17, предложенного Л.Г. Перетцем в 1930 году [4].
Далее было проведено полногеномное секвенирование генома штамма и установлены его основные характеристики (табл. 1).
Таблица 1
Основные характеристики генома E. coli M-17
|
Характеристики |
E. coli M-17 |
|
Размер генома, п.н. |
4.483.110 |
|
GC, % |
50.7 |
|
Количество генов |
5.107 |
|
Количество псевдогенов |
80 |
|
CDS (последовательности, кодирующие белки) |
4.930 |
|
Количество представленных подсистем согласно RAST |
595 |
|
CRISPR-регион |
1 |
|
Опероны синтеза бактериоцинов: V M B |
1 (KLD50841.1, KLD50842.1) 1 (KLD42107.1) 1 (KLD42108.1) |
|
rRNA |
13 |
|
tRNA |
77 |
Очевидно, что данный штамм имеет «кишечное» происхождение, так как в его геноме присутствует большое количество псевдогенов и компонент системы устойчивости к солям желчи - мембранный протеин DamX (KLD50520.1).
В геноме обнаружены опероны синтеза колицина V, М и В – бактериоцинов, характерных для E. coli, которые обуславливают высокий уровень антагонистической активности данного штамма.
С использованием программы CrisprFinder проанализирован CRISPR-регион изучаемого штамма, расположенный в пределах 57 контига (LBDD01000002). Установлено, что по структурной организации CRISPR-регион исследуемого штамма отнесится к I-E типу, что типично для представителей рода Escherihia [14]. Уникальные последовательности спейсеров и повторов данного региона можно рассматривать как «метку штамма» и впоследствии использовать для его индикации (рис. 2).
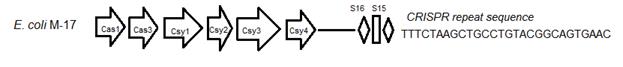
Рис. 2. Организация CRISPR-кассеты E. coli М-17. Стрелками обозначены соответствующие Cas-белки, ромбы обозначают повторы, прямоугольники – уникальные спейсеры
Присутствие в составе генома CRISPR-региона говорит о внедрении фагов в прошлом. О том, что изучаемый микроорганизм подвергался атакам фагов, свидетельствует и тот факт, что в его геноме обнаружены профаги - гены, кодирующие фаговые белки, элементы капсида, ферменты репликации, гены-активаторы белков фага - всего 96 детерминант.
Метаболизм штамма изучен с использованием RAST. Установлено, что в геноме детерминировано несколько путей центрального метаболизма углеводов: гликолиз, путь Энтнера-Дудорова и пентозофосфатный путь; выявлено, что данные метаболические пути штамма не имеют особенностей. В целом E.coli M-17 обладает выраженным метаболическим потенциалом и способен утилизировать широкий спектр субстратов.
С использованием RAST и программы ResFinder полногеномная последовательность штамма была проанализирована на наличие генов антибиотикорезистентности. Установлено, что в состав генома изучаемого штамма входит кластер транспортных генов (mdtABCD), наличие которого обуславливает устойчивость к новобиоцину и доксихолату [7]. Также в геноме представлены гены оперона, регулирующего экспрессию генов, отвечающих за систему эффлюкса лекарственных, в том числе и антибактериальных препаратов (MAR локус), а также другие виды молекулярных эффлюксных помп - CmeA, CmeB, TolC, AcrR MacA, MacB, а также эффлюксные помпы семейств MATE и MFS. Все перечисленные детерминанты имеют хромосомную локализацию, типичны для представителей рода и не представляют угрозы в плане трансмиссивного распространения [6].
Далее геном был проанализирован с использованием программ PlasmidFinder-1.3, VirulenceFinder-1.5 и PathogenFinder. В результате установлено, что исследуемый штамм не содержит интегрированных плазмид, детерминант вирулентности и патогенности.
На современном этапе существует ряд методов, позволяющих типировать штаммы с использованием их полногеномных последовательностей. Наиболее распространенным является метод мультилокусного сиквенс типирования (MLST), предполагающий установление аллельных профилей отдельных «генов домашнего хозяйства» штамма [13]. Для проведения MLST штамма использовали программу MLST-1.8. В данной схеме анализируются фрагменты семи «генов домашнего хозяйства» – аденилаткиназы (adk), фумарат гидратазы (fumC), ДНК гиразы (gyrB), изоцитрат дегидрогеназы (icd), малат дегидрогеназы (mdh), аденилосукцинат дегидрогеназы (purA) и рекомбиназы А (recA). Обнаружено, что исследуемый штамм E. coli M-17 принадлежит к 141 сиквенс-типу (ST-141) (табл. 2).
Таблица 2
Аллельные профили «генов домашнего хозяйства» исследуемого штамма
|
Штамм |
Аллели |
ST |
||||||
|
adk |
fumC |
gyrB |
icd |
mdh |
purA |
recA |
||
|
E. coli M-17 |
13 |
52 |
10 |
14 |
17 |
25 |
17 |
141 |
Согласно данным научной литературы микроорганизмы с таким сиквенс-типом ранее не вызывали эпидемических вспышек заболеваний [3]. Данные, представленные в международной базе данных [15], указывают на то, что сиквенс-тип характерен для непатогенных эшерихий человеческого происхождения.
С использованием программного продукта SerotypeFinder-1.1. определен серотип штамма. Эта программа использует полногеномные последовательности и позволяет анализировать детерминанты антигенов микроорганизма. Для E. coli это ген, кодирующий структурный белок флагеллин fliC (KLD44527.1) и белки O-антигена - Wzx (KLD49178.1) и Wzy (KLD49266.1). Установлено, что штамм E. coli M-17 имеет серотип О2:Н6, который соответствует серотипу E. coli M-17 согласно «Регламенту производства колибактерина сухого» [5].
Заключение. В результате проведенной работы установлено, что штамм E. coli M-17 (ФГУП «НПО «Микроген») обладает типичными биохимическими свойствами, заявленными в ОФС на колисодержащие пробиотики. Для штамма характерны следующие массы ионизированных белков: 2329, 2569, 2940, 3168, 3638, 5340, 6243, 6255, 6276, 8875, 9713. При анализе полногеномной последовательности у штамма не обнаружены трансмиссивные гены антибиотикорезистентности, гены патогенности, вирулентности и интегрированные плазмиды. В геноме детерминирован ряд фаговых генов - гены, кодирующие фаговые белки, элементы капсида, ферменты репликации, гены-активаторы белков фага - всего 96 детерминант. Определен серотип штамма – О2:Н6, который соответствует серотипу E. coli M-17 согласно Регламенту производства колибактерина сухого (1980). Впервые с использованием метода мультилокусного сиквенс-типирования был установлен сиквенс-тип штамма - ST141. Полногеномная последовательность генома штамма E. coli M-17 (ФГУП «НПО «Микроген») задепонирована в международной базе данных GenBank под соответствующим номером проекта - LBDD00000000.1.
Библиографическая ссылка
Белова И.В., Точилина А.Г., Соловьева И.В., Горлова И.С., Ефимов Е.И., Жирнов В.А., Иванова Т.П. ФЕНОТИПИЧЕСКИЕ И ГЕНОТИПИЧЕСКИЕ ХАРАКТЕРИСТИКИ ПРОБИОТИЧЕСКОГО ШТАММА E. COLI M-17 // Современные проблемы науки и образования. 2017. № 3. ;URL: https://science-education.ru/ru/article/view?id=26533 (дата обращения: 05.02.2026).
DOI: https://doi.org/10.17513/spno.26533



