Многие белки, в частности GFP-подобные флуоресцирующие белки, могут быть подвергнуты химической модификации, например ацилированию янтарным ангидридом[3]. Однако в белке может присутствовать несколько, до нескольких десятков лизинов[4]. И даже при соотношении белок/модифицированная аминогруппа 1/1 после ацилирования возникает вопрос, какие именно остатки лизина были модифицированы поскольку это может сильно повлиять на взаимодействие GFP-подобного белка с другими белками.
Цель исследования
Целью данной работы является на примере кораллового флуоресцирующего белка SAASoti [статья находится в печати] описать метод определения положения модифицированных ацилированием боковых групп лизинов. Для идентификации аминокислот используются метод масс-спектрометрии MALDI TOF/TOF.
Материалы и методы
Масс-cпектрометрия
После проведения реакции модификации доступных янтарному ангидриду остатков лизина белка, образцы были очищены на обращенной фазе C18 P10. Для этого к 10 мкл пробы добавляли по 10 мкл 0.1% ТФУ, промывку проводили 5 объемами (по 10 мкл) 0.1% ТФУ, а элюцию - 10 мкл 60% ацетонитрила в 0.1% ТФУ. Далее пробы были подсушены и подвергнуты протеолизу трипсином, для чего к ним добавили по 10 мкл раствора модифицированного трипсина (Promega) в 0.05М NH4HCO3 с концентрацией 10 мкг/мл. Гидролиз проводили в течение 5 ч при 37°С, затем остановили протеолиз добавлением 20 мкл 0.5 % ТФУ в 10 % растворе водного ацетонитрила. Этот раствор использовали для получения MALDI-масс-спектров. На масс-спектрометрической мишени смешивали по 1 мкл раствора образца и 0.5 мкл раствора 2,5-дигидроксибензойной кислоты (Aldrich, 20 мг/мл в 20 % водном ацетонитриле, 0.5% ТФУ), полученную смесь высушивали на воздухе.
Масс-спектры были получены на MALDI-времяпролетном масс-спектрометре Ultraflextreme BRUKER (Германия), оснащенном УФ лазером (Nd) в режиме положительных ионов с использованием рефлектрона; точность измеренных моноизотопных масс после докалибровки по пикам автолиза трипсина составляла 0.005 % (50ррм). Спектры получали в диапазоне масс 600-5000 m/z, выбирая мощность лазера, оптимальную для достижения наилучшего разрешения. Для получения спектров фрагментации использовали тандемный режим прибора, точность измерения фрагментных ионов была не хуже 1 Да.
Соотнесение экспериментально полученных и теоретически рассчитанных масс триптических пептидов белка SAASoti осуществляли при помощи программы Mascot [5]. Масс-спектры были обработаны с помощью программного пакета FlexAnalysis 3.3 (Bruker Daltonics, Германия), созданы пик-листы htm формата. При помощи программы Mascot (опция «пептидный фингерпринт) провели формальный поиск в домашней базе данных, куда предварительно были внесены последовательности искомого белка в полноразмерной и процессированной форме. Поиск проводили с указанной выше точностью, с учетом возможных модификаций остатков лизина и аминогруппы N-конца белка янтарным ангидридом, образования гетероцикла в сайте флуорофора и возможного окисления метиониновых остатков кислородом воздуха. Тип протеолиза указывали триптический, но предполагали значительное (до 5-ти подряд) количество пропусков в работе фермента из-за внесенных на остатки лизина SAASoti модификаций. Для подтверждения наличия либо отсутствия модификаций янтарным ангидридом определенных остатков лизина были получены спектры фрагментации отдельных пептидов. С использованием программного обеспечения Biotools 3.2 (Bruker Daltonics, Германия) проведен дополнительный анализ данных по объединенным MS+MS/MS результатам.
Результаты и обсуждение
Для того, чтобы установить, какие именно из ε-аминогрупп модифицированы, аликвота с модифицированным SAASoti была трипсинизирована. Триптические пептиды, полученные в результате гидролиза, были проанализированы на MALDI TOF/TOF. Трипсин расщепляет полипептидную цепь после остатков лизина и аргинина. В случае модификации лизина, вместо аминогруппы, боковая цепь лизина будет оканчиваться карбоксильной группой. Трипсин не может расщепить цепь после такого остатка из-за стерических затруднений [6]. В случае модификации ε-аминогруппы янтарным ангидридом, лизин получает добавку к весу 100,7 Да.
В полученном наборе триптических пептидов с помощью MALDI удалось расшифровать 29 пиков масс. Последовательности расшифрованных триптических пептидов представлены на рисунке 1.
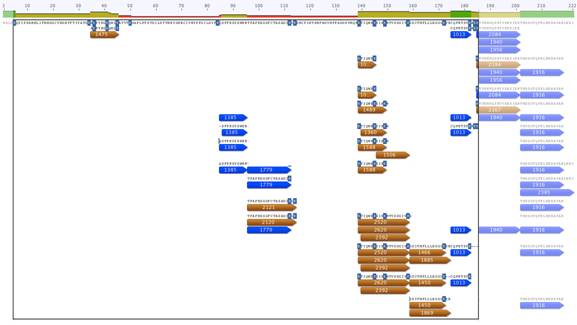
Рисунок 1. Карта расположения пиков масс триптических пептидов относительно последовательности белка SAASoti. В верхней строке отображена последовательность исходного белка. Лизины контрастированы синим фоном. Триптические пептиды отображены синими и оранжевыми полосками с подписанными на них массами пика в Дальтонах. Массы пиков, отображенных синими полосками, совпадают с расчетными массами. Массы пиков, отображенных оранжевыми полосками, отличаются от расчетных на массу кратную 100,7 Да.
Расшифрованные триптические пептиды содержат 47% последовательности SAASoti, что можно наблюдать на рисунке 2. Однако лизины в последовательности SAASoti распределены неравномерно и 12 из 17 лизинов оказались в триптических пептидах, расшифрованных на МАЛДИ.
В наборе расшифрованных пиков были выделены те, которые имеют прибавку к массе кратную 100,7 Да к теоретической совокупной массе аминокислот. Массы этих пиков также присутствуют в таблице 1. Всего было получено 7 пиков с единичной модификацией лизина, 4 пика с двойной модификацией и 1 с тройной модификацией.

Рисунок 2. Аминокислотная последовательность белка SAASoti. Жирным шрифтом отмечены лизины. Серым фоном отмечены части последовательности расшифрованной с помощью МАЛДИ
Сравнение полученного набора масс с теоретическими весами триптических пептидов позволяет утверждать, что модификации подверглись остатки лизина 36, 41, 112, 139, 145, 149, 172 и 185.
Выводы
Представленный метод идентификации модификации боковой цепи аминокислоты с помощью масс-спектрометра MALDI TOF/TOF может быть использован для практических задач.
Работа выполнена при поддержке ЦКП «Прикладные биотехнологии», идентификатор проекта RFMEFI62114X0002.
Рецензенты:Курочкин И.Н., д.х.н., профессор, зав. Лабораторией Экобиокатализа кафедры Химической Энзимологии Химического факультета Московского Государственного Университета имени М.В. Ломоносова, г.Москва;
Капрельянц А.С., д.б.н., зав. Лабораторией стрессов микроорганизмов института Биохимии им. А.Н. Баха РАН, г.Москва.
Библиографическая ссылка
Лапшин Г.Д., Винокуров Л.М., Савицкий А.П. ИДЕНТИФИКАЦИЯ ХИМИЧЕСКИ МОДИФИЦИРОВАННЫХ АМИНОКИCЛОТНЫХ ОСТАТКОВ МЕТОДОМ МАЛДИ // Современные проблемы науки и образования. 2014. № 6. ;URL: https://science-education.ru/ru/article/view?id=16365 (дата обращения: 06.01.2026).



