Введение
Использование сухих пятен крови (СПК) в качестве клинических образцов для проведения молекулярно-биологических исследований получило свое распространение в 60‑х годах ХХ века и с тех пор является одним из надёжных инструментов в клинической лабораторной диагностике. По сравнению с забором крови из вены получение капли крови является менее инвазивным методом и требует минимального объёма крови – 30-50 мкл, что особенно важно при работе с новорождёнными, детьми и пациентами с анемией. При этом такие образцы обладают достаточно высокой стабильностью, даже в течение нескольких лет, и не требуют особого температурного режима при хранении и транспортировке, что играет значительную роль при молекулярно-генетических исследованиях в отдаленных и труднодоступных регионах [1-3].
В настоящее время применение сухих пятен крови охватывает широкий спектр различных направлений, таких как неонатальный скрининг (выявление наследственных заболеваний), молекулярно-генетические исследования инфекционных заболеваний (ВИЧ, гепатит). В судебно-медицинской экспертизе они используются при идентификации личности, установлении отцовства, генетическом профилировании. В фармакогенетике ДНК, полученная из сухих пятен крови, позволяет определять генетические особенности пациента, оказывающие влияние на метаболизм лекарственных препаратов, что позволяет персонифицировать лечение, достигать терапевтического эффекта в короткие сроки, а также избегать побочных эффектов. Значительную роль использование сухих пятен крови играет также при создании биобанков, так как не требует огромного количества морозильных камер, занимает достаточно мало пространства, а отсутствие требований к транспортировке позволяет получать образцы из удаленных регионов [4-6].
Одним из основных типов матриц, на которые наносятся пятна крови, являются целлюлозно-бумажные FTA-карты, представляющие собой специализированный носитель, пропитанный химическими веществами (детергентами, хелатирующими агентами и буферами), которые обеспечивают лизис клеток, стабилизацию и иммобилизация нуклеиновых кислот и защиту от контаминации. В результате нанесения капли крови на матрицу молекула ДНК оказывается защищенной от различного рода воздействий, таких как УФ-излучение, влажность, перепады температуры, а также от воздействий ферментов и бактериального роста [6; 7].
Процесс выделения ДНК из сухих пятен крови включает четыре основных этапа – десорбция образца с матрицы, лизис клеток и высвобождение ДНК, очистка ДНК от примесей (белков, полисахаридов и др.), концентрация и элюция ДНК. Эффективность каждого этапа может значительно варьировать в зависимости от типа матрицы и используемого метода экстракции. Несмотря на то, что большинство методов экстракции подходит для проведения реакции ПЦР, для устранения ингибирования может требоваться оптимизация условий различных этапов выделения из сухих пятен крови [8].
Так, в ряде работ рассмотрены различные проблемы, возникающие в ходе экстракции ДНК из сухих пятен крови, а именно ухудшение качества образца вследствие недостаточной отмывки, недостаточного количества клеток после десорбции образца с матрицы и ряд других [9; 10].
В разных источниках литературы показано несколько вариантов оптимизаций и модификаций этапов выделения ДНК. К наиболее часто встречающимся относят: увеличение количества дисков (при необходимости получения большого количества ДНК для полногеномного секвенирования), добавление этапа предварительной инкубации диска в небольшом объеме буфера (ТЕ-буфер, вода, элюирующий буфер) с нагреванием или без нагревания, уменьшение объема сорбента, различных буферов экстракции [11-13].
Цель работы – оптимизация протокола сорбционного метода выделения ДНК из сухих пятен крови.
Материалы и методы исследования
В исследовании использовали образцы (N=30) цельной периферической крови, полученной методом флеботомии в пробирках с ЭДТА. Процедура забора крови осуществлялась у сотрудников ФГБУ «ЦСП» ФМБА России после добровольного согласия.
Подготовка образцов.
Для приготовления сухих пятен крови был использован ДНК-архив («Алкор-Био», Россия) на основе бумажно-целлюлозного волокна.
Для подготовки образцов сухих пятен крови проводили следующие этапы: нанесение капли крови в объёме 50 мкл на каждую матрицу, высушивание в течение 24 часов при комнатной температуре в защищенном от солнечных лучей месте.
Экстракция ДНК.
Экстракция ДНК из образцов сухих пятен крови проводилась набором «ДНК-Сорб» (производство ФГБУ «ЦСП» ФМБА России) согласно инструкции производителя и включала в себя несколько этапов: лизис клеток, преципитацию, три последовательные процедуры отмывки и элюирование с помощью ТЕ-буфера. Элюция проводилась в двух объёмах – 35 и 50 мкл.
Для экстракции ДНК из сухих пятен крови предварительно получали панчи диаметром 6 мм, маркировали и помещали каждый в пробирку 1,5 мл типа Эппендорф. Выделение ДНК проводили несколькими протоколами:
1 протокол – выделение согласно стандартному протоколу (СП).
В пробирки с панчами добавляли 100 мкл воды, перемешивали на вортексе и кратко центрифугировали для сброса капель с крышки. Затем добавляли 300 мкл лизирующего буфера и ставили в термостат для инкубации на 5 минут при 65 °С, периодически помешивая. После инкубации пробирки кратковременно центрифугировали для осаждения капель, затем аккуратно переносили надосадочную жидкость, не задевая диск, в новую пробирку. В пробирки добавляли сорбент объёмом 25 мкл и инкубировали при комнатной температуре в течение 7 минут, периодически помешивая. Дальнейшее выделение ДНК проводили согласно инструкции производителя.
2 протокол - стандартный протокол с 1 отмывкой (СП-1).
В пробирки с панчами добавляли 100 мкл воды, перемешивали на вортексе и кратко центрифугировали для сброса капель с крышки. Затем добавляли 300 мкл лизирующего буфера и ставили в термостат для инкубации на 5 минут при 65 °С, периодически помешивая. После инкубации пробирки кратковременно центрифугировали для осаждения капель, затем аккуратно переносили надосадочную жидкость, не задевая диск, в новую пробирку. В пробирки добавляли сорбент объёмом 25 мкл и инкубировали при комнатной температуре в течение 7 минут, периодически помешивая. После инкубирования проводили центрифугирование пробирок и отбирали надосадочную жидкость. С осаждённым сорбентом проводили первую отмывку согласно инструкции, вторая отмывка проводилась только однократно.
3 протокол - протокол выделения с использованием уменьшенного объёма лизирующего буфера и сорбента (СП/2).
В пробирки с панчами добавляли лизирующий буфер в объёме 150 мкл, пробирки перемешивали на вортексе и сбрасывали капли центрифугированием. После этого образцы инкубировали в термостате в течение 5 мин. при температуре 65 °С, периодически помешивая. После инкубации пробирки кратковременно центрифугировали для осаждения капель, затем аккуратно переносили надосадочную жидкость, не задевая диск, в новую пробирку. В пробирки добавляли сорбент объёмом 12,5 мкл и инкубировали при комнатной температуре в течение 7 минут, периодически помешивая. Дальнейшее выделение ДНК проводили согласно инструкции производителя.
4 протокол - предварительная инкубация СПК в лизирующем буфере в течение 30 минут при температуре 56 °С (СП+30̓ ЛБ).
В пробирки с панчами добавляли 300 мкл воды, перемешивали на вортексе и кратко центрифугировали для сброса капель с крышки. Затем ставили в термостат для инкубации на 5 минут при 65 °С, периодически помешивая. После инкубации пробирки кратковременно центрифугировали для осаждения капель, затем аккуратно переносили надосадочную жидкость, не задевая диски, в новую пробирку. Затем добавили сорбент объёмом 25 мкл и инкубировали при комнатной температуре в течение 7 минут, периодически помешивая. Дальнейшее выделение ДНК проводили согласно инструкции производителя.
5 протокол - предварительная инкубация СПК в воде в течение 20 минут при комнатной температуре (СП + 20̓ вода).
В пробирки с дисками добавляли 100 мкл деионизированной воды, после чего все пробирки кратковременно перемешивались на вортексе и инкубировались в течение 20 минут при комнатной температуре. Во время инкубации пробирки периодически встряхивались на вортексе. Панчи при этом всегда должны были быть погружены в раствор. После инкубации добавлялся лизирующий буфер и проводилось выделение согласно стандартному протоколу, описанному выше.
6 протокол - предварительная инкубация СПК в ТЕ-буфере в течение 20 мин. при комнатной температуре (СП+ 20̓ ТЕ).
В пробирки с дисками добавляли ТЕ-буфер объемом 100 мкл, после чего кратковременно перемешивали на вортексе и инкубировали в течение 20 минут при комнатной температуре. Во время инкубации пробирки периодически встряхивали на вортексе. Панчи при этом всегда должны быть погружены в раствор. После инкубации в пробирки добавлялся лизирующий буфер и проводили выделение согласно стандартному протоколу.
В качестве набора сравнения был использован оптимизированный протокол для выделения ДНК из сухих пятен крови к набору DNeasy Blood & Tissue Kit (Qiagen, США). Согласно инструкции производителя, основное отличие от стандартного протокола – длительная инкубация в буфере сначала 15 минут при температуре 90 °С, а затем 60 минут при температуре 56 °С.
Концентрацию выделенной ДНК измеряли спектрофотометрическим и флуориметрическим методами с использованием спектрофотометра/флуориметра DeNovix DS-11 FX (DeNovix Inc., США). Каждый образец был измерен в трёх повторах при комнатной температуре.
Оценку качества полученной ДНК проводили с помощью ПЦР-РВ. Праймеры и флуоресцентный зонд были подобраны на ген домашнего хозяйства ABL1 с помощью программы Primer3, синтез был проведен в АО «ГенTерра» (Россия). Смесь для ПЦР-РВ содержала все необходимые компоненты, а именно: смесь праймеров и зондов – 10 мкл, ПЦР-буфер – 5 мкл, dNTP – 0,5 мкл, Taq-полимераза – 0,5 мкл (производство ЦСП ФМБА, Россия).
Реакция амплификации проводилась с использованием амплификатора CFX 96 (BioRad, США) по следующей программе: 95 °С – 15 мин., затем 45 циклов – 95 °С 15 сек., 60°С – 60 сек. Детекция флуоресцентного сигнала осуществлялась по каналу HEX. Оценка полученных сигналов проводилась с учетом корректного прохождения положительных и отрицательных контролей. В качестве положительного контроля использовалась плазмида с искусственно синтезированной вставкой, в качестве отрицательного контроля – деионизованная вода. Для повышения точности и надежности результатов каждый образец был исследован в трёх повторах.
Статистическая обработка данных проводилась с использованием программного обеспечения Microsoft Exсel 2010 (США) и StatTech v.4.7 (разработчик ООО «СтатТех», Россия). Различия считались значимыми при p<0,05.
Результаты исследования и их обсуждение
Концентрация ДНК, измеренная спектрофотометрическим и флуориметрическим методами, коэффициенты чистоты А260/280 и А260/230, показатели Ct представлены в таблице 1.
Таблица 1
Сравнительные результаты полученных данных
|
Метод |
1 протокол (СП) |
2 протокол (СП-1) |
3 протокол (СП/2) |
4 протокол (СП+ 30̓ ЛБ) |
5 протокол (СП+ 20̓ вода) |
6 протокол (СП+ 20̓ ТЕ) |
|
Концентрация ДНК (спектрофотометрия), нг/мкл |
34,24±1,96 |
34,46±3,29 |
23,92±2,11 |
27,32±0,95 |
8,03±0,86 |
16,10±1,48 |
|
Концентрация ДНК (флуориметрия), нг/мкл |
1,63±0,28 |
1,75±0,14 |
1,25±0,20 |
0,56±0,03 |
0,15±0,02 |
0,09±0,04 |
|
Спектральные характеристики A260/280 |
1,27±0,03 |
1,92±0,12 |
1,81±0,04 |
1,77±0,03 |
2,13±0,09 |
1,75±0,05 |
|
Значение Ct состандартным отклонением |
26,95±1,41 |
26,85±0,94 |
28,41±1,35 |
28,70±0,42 |
36,75±1,37 |
32,77±0,72 |
Таблица составлена авторами на основе собственных данных.
При выделении ДНК из СПК наиболее высокие результаты показали протокол 1 (выделение по стандартному протоколу) 34,24±1,96 нг/мкл (ДИ 95%: 30,4 – 38,07) и протокол 2 (выделение с 1 отмывкой раствора 2) 34,46±3,29 нг/мкл (ДИ 95%: 28,01– 40,9). Снижение концентрации ДНК почти в 1,5 раза было отмечено при использовании протокола 3 (уменьшение лизирующего буфера и сорбента в два раза) 23,92±2,11 нг/мкл (ДИ 95%: 19,79 – 28,04) и протокола 4 (с предварительной инкубацией в ЛБ при 56 °С в течение 30 минут) 27,32±0,95 нг/мкл (ДИ 95%: 25,36 – 29,09).
Использование протоколов 5 (предварительная инкубация в воде 20 минут при комнатной температуре) и 6 (предварительная инкубация в элюенте 20 минут при комнатной температуре) показали наиболее низкие значения концентрации, а именно: 8,03±0,86 нг/мкл (ДИ 95%: 6,35 – 9,71) и 16,10±1,48 нг/мкл (ДИ 95%: 13,10 – 18,9) соответственно.
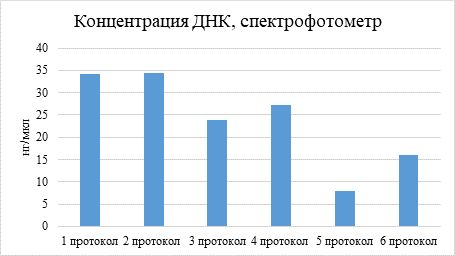
Рис. 1. Значения концентрации ДНК, выделенной разными протоколами (составлено авторами по результатам данного исследования)
Результаты анализа спектральных характеристик А260/280 показали высокий уровень чистоты ДНК (рис. 2).

Рис. 2. Значения спектральных характеристик ДНК, выделенной разными методами (составлено авторами по результатам данного исследования)
Наиболее оптимальный уровень наблюдался при использовании протокола 2 – 1,92±0,12 (ДИ 95%: 1,69 – 2,15). При использовании протокола 5 был получен повышенный уровень коэффициента А260/280 - 2,13±0,09 (ДИ 95%:1,94 – 2,30), что говорит о возможном загрязнении белками. Выделение ДНК из СПК протоколами 3, 4, 6 показало одинаковый уровень характеристики А260/280 1,81±0,04 (ДИ 95%:1,7 – 1,8), 1,77±0,03 (ДИ 95%:1,74 –1,83), 1,77±0,05 (ДИ 95%: 1,67 – 1,86) соответственно. Самые низкие значения были получены при использовании протокола 1 – 1,27±0,03 (ДИ 95%:1,2 – 1,3).
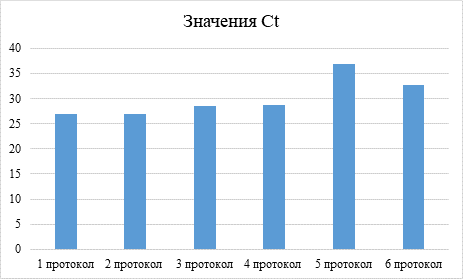
Рис. 3. Показатели Ct выделенной ДНК разными протоколами (составлено авторами по результатам данного исследования)
При постановке ПЦР наиболее ранние выходы получены на образцах, выделенных 1 протоколом, Ct составляет 26,95±1,41 (ДИ 95%: 26,53 – 27,36) и 2 протоколом – 26,85±0,94 (ДИ 95%:26,53 – 27,36). Поздние значения показали протоколы 5 и 6 – 36,75±1,37 (ДИ 95%:36,3 – 37,14) и 32,77±0,72 (ДИ 95%:32,56 – 32,98) соответственно (рис. 3).
По итогам проведенных экспериментальных работ были выбраны наиболее оптимальные методы выделения – 1, 2 и 3 протоколы.
Для дальнейшей оптимизации проведено выделение ДНК с увеличением объёма элюирующего буфера до 50 мкл, а также параллельное выделение набором сравнения DNeasy Blood & Tissue Kit согласно оптимизированному протоколу производителя. Полученные данные представлены в таблице 2.
Таблица 2
Сравнительные результаты полученных данных с увеличением объёма элюции
|
Показатели |
1 протокол (СП) |
2 протокол (СП-1) |
3 протокол (СП/2) |
Qiagen |
||||
|
35 мкл |
50 мкл |
35 мкл |
50 мкл |
35 мкл |
50 мкл |
35 мкл |
50 мкл |
|
|
Концентрация ДНК (спектрофотометрия), нг/мкл |
34,24± 1,96 |
65,34± 7,41 |
34,46± 3,29 |
74,94± 5,50 |
23,92± 2,11 |
87,8± 6,1 |
3,55± 0,22 |
2,10± 0,13 |
|
Концентрация ДНК (флуориметрия), нг/мкл |
1,63± 0,28 |
1,99± 0,18 |
1,75± 0,14 |
2,66± 0,23 |
1,25± 0,20 |
1,4± 0,1 |
1,56± 0,08 |
0,99± 0,07 |
|
Спектральные характеристики A260/280 |
1,27± 0,03 |
1,55± 0,02 |
1,92± 0,12 |
1,53± 0,01 |
1,81± 0,04 |
1,5± 0,01 |
1,32± 0,92 |
1,26± 0,04 |
|
Значение Ct со стандартным отклонением |
26,95± 1,41 |
28,24± 1,39 |
26,85± 0,94 |
27,73± 0,73 |
28,41± 1,35 |
27,6± 0,9 |
30,98± 0,67 |
30,77± 0,10 |
Таблица составлена авторами на основе собственных данных.
Увеличение объема элюирующего буфера до 50 мкл показало незначительное снижение уровня выхода ДНК и значения Сt составили – 28,24±1,39 (ДИ 95%: 27,83 – 28,64), 27,73±0,73 (ДИ 95%: 27,51 – 27,94), 28,41±1,35 (ДИ 95%: 27,40 – 27,90) в 1, 2, 3 методах соответственно. При этом наблюдалось увеличение таких показателей, как концентрация, измеренная спектрофотометрическим и флуорометрическим методами, а также спектральная характеристика 260/280 (табл. 2).
Применение t-критерия Стьюдента в анализе данных составило более 0,05 для всех исследуемых показателей, что свидетельствует о статистической недостоверности различий между анализируемыми оптимизированными протоколами выделения ДНК из сухих пятен крови.
Использование ДНК, полученной при выделении из сухих пятен крови, является важной составной частью молекулярно-биологического исследования, от которой зависит получение валидных результатов. Применение FTA-карт имеет ряд преимуществ перед использованием цельной крови: малоинвазивность забора материала, способность сохранять ДНК при длительном хранении в комнатной температуре (в течение нескольких лет), удобство транспортирования (не занимает много места и не требует специальных температурных условий), а также возможность использования 1 образца несколько раз, не подвергая его многократным циклам заморозки/разморозки.
В настоящее время существует множество различных протоколов выделения ДНК из сухих пятен крови с помощью разных методов.
В исследовании (2017) Горбенко А.С. описывал оптимизированную процедуру выделения ДНК с использованием сорбционного метода, в ходе которого к сухим пятнам крови, помещенным в пробирку, был добавлен лизирующий раствор, а затем проводилось инкубирование при температуре 65 °С в течение 1 часа. Сравнительный анализ выделения из цельной крови и сухих пятен крови показал высокую корреляцию между образцами цельной крови и сухих пятен [14].
Седых А.В. и др. (2023) провели сравнение различных наборов для выделения ДНК из сухих пятен крови (с использованием магнитной силики и метода спирового осаждения НК) по следующим критериям: эффективность анализа ПЦР, чистота ДНК (А260/280), концентрация ДНК, измеренная спектрофотометрическим методом, а также время анализа и количество этапов, входящих в протокол. В ходе экспериментов было продемонстрировано, что все четыре набора реагентов показали достаточно высокие уровни выбранных критериев и могут применяться на практике в молекулярно-биологических исследованиях [15].
Van Biesen N. et al. (2025) проводили сравнение пяти различных методов выделения ДНК из образцов сухих пятен крови – три различных метода выделения на основе колонок и два метода на основе нагревания с использованием буфера ТЕ и с использованием смолы Chelex-100. Была показана низкая концентрация ДНК при использовании колоночного метода выделения (2,10 нг/мкл (ДИ 95%:1,67 − 2,53)), показатель чистоты 260/280 составил 1,72 (ДИ 95%: 1,37 – 2,07) и 260/230 - 0,33 (ДИ 95%: 0,26 – 0,39), что подтверждается полученными результатами (табл. 2) [8].
В ходе экспериментов по оптимизации объёма элюирующего буфера было продемонстрировано, что использование колоночного метода с меньшим объёмом элюирования увеличивает концентрацию выделенной ДНК по сравнению с другими методами, что также демонстрируют эксперименты, проведённые Van Biesen N. [8].
Kumar A. et al. в 2019 году проводили оптимизацию экстракции ДНК с помощью предварительной инкубации в растворе PBS (200 мкл) в течение ночи при комнатной температуре, последующей экстракцией с использованием колоночного метода (QIAamp DNA, Qiagen) и с помощью набора реагентов на магнитных частицах (ChargeSwitch Forensic DNA Purification Kit, Invitrogen). В результате проведенных экспериментов не наблюдалось увеличения концентрации геномной ДНК и при этом был продемонстрирован большой разброс в значениях концентрации (64,8-720 нг) и спектральной характеристики 260/280 – 1,8-2,9 [16].
Выбранный авторами протокол выделения ДНК основан на сорбционном методе, так как он достаточно широко применяется в клинической практике благодаря своей эффективности и простоте. Он включает в себя следующие этапы: лизис клеток, во время которого происходит разрушение клеточных мембран и высвобождение ДНК, связывание сорбента и ДНК, отмывка связанной с сорбентом ДНК от различных примесей – белков, липидов и других веществ, элюция в ТЕ-буфере (отделение ДНК от сорбента). Протоколы оптимизации включали в себя такие изменения, как предварительная инкубация в различных растворах, изменение объема лизирующего буфера и сорбента, удаление одной отмывки, а также изменение объема элюирующего буфера.
Заключение
Метод выделения ДНК из сухих пятен крови на картах FTA является надёжным, простым, безопасным и экономичным инструментом для сбора, хранении и анализа ДНК. Его ключевые преимущества, а именно: стабильность образцов, инактивация патогенов и простота выделения, обеспечили ему широкое применение в научных исследованиях и практических областях, от криминалистики до клинической диагностики. Хотя метод имеет некоторые ограничения (количество ДНК, фрагментация, потенциальные ингибиторы), возможность оптимизации протоколов выделения, включая автоматизацию и адаптацию для NGS, расширяет сферы его использования.
Полученные в ходе экспериментов результаты показывают незначительные расхождения в анализируемых показателях, что говорит о возможности использования данного метода для выделения ДНК из сухих пятен крови без оптимизации протокола.
Карты FTA остаются незаменимым решением для задач, требующих надежного сбора, транспортировки, долгосрочного хранения и быстрого получения пригодной для ПЦР ДНК из образцов цельной крови.



