Во всем мире в 2018 году было зарегистрировано более 528 000 новых случаев и 266 000 летальных исходов от инвазивного рака шейки матки (ИРШМ) [1]. Инфекция вирусом папилломы человека (ВПЧ, HPV) является неотъемлемой причиной ИРШМ [2]. Несмотря на то что подавляющее большинство инфекций ВПЧ проходит спонтанно [3], персистирование ВПЧ-инфекции может приводить к интраэпителиальным поражениям, которые гистологически классифицируются как цервикальная интраэпителиальная неоплазия (ЦИН) 1–3 степени. Около 1/3 CIN3 прогрессирует до инвазии через 30 лет [4]. CIN1 считается морфологическим отражением инфекции ВПЧ, а CIN2 представляет собой смесь CIN1 и CIN3, часто регрессирующую [5]. Механизмы канцерогенеза изучены лишь частично. Прогрессирование до интраэпителиальных предраковых заболеваний происходит из-за нарушения в сигнальных путях, регулирующих клеточный цикл, опосредованных избыточной экспрессией вирусных белков E6 и E7, которые функционально инактивируют продукты генов RB и p53. Прогрессия до уровня инвазии включает в себя накопление генетических ошибок. Весь процесс является многоэтапным и включает ряд генетических и эпигенетических изменений [6].
Скрининг, основанный на ВПЧ-тестировании более эффективен, чем цитологический [7]. Однако положительная прогностическая ценность теста на ВПЧ для ЦИН высокой степени злокачественности низкая. Цитологическая верификация эффективна [7], но влечет за собой высокую частоту кольпоскопии и повторных тестов [8]. Поэтому новые маркеры, позволяющие повысить эффективность скрининга, были бы очень полезны.
МикроРНК (miRNAs) - это короткие некодирующие РНК, которые регулируют экспрессию генов, катализируя разрушение мРНК либо ингибируя трансляцию мРНК в белок [9]. Зрелая miRNA представляет собой одноцепочечную ~ 22 нуклеотидов РНК, получающуюся из первичного транскрипта [10]. miRNAs являются эпигенетическими регуляторами, специфичны для определенной ткани и модулируют экспрессию генов путем взаимодействия с комплементарными нуклеотидными последовательностями мРНК-мишеней [11]. miRNAs могут быть сверхэкспрессированы или гипоэкспрессированы при раке [12], что связано с генетическими и эпигенетическими изменениями [13; 14].
Данные по клеточным линиям и предраковым заболеваниям подтверждают участие miRNAs в каждом этапе развития ИРШМ [15; 16]. Тем не менее пока мало что известно о специфических микроРНК, задействованных в этом, и механизмах их дерегуляции [17]. Аберрантная экспрессия miRNA, по-видимому, является результатом HPV-инфекции. Некоторые гены miRNA локализуются в сайтах, где происходит интеграция HPV ДНК. Белки, кодируемые HPV, могут влиять на экспрессию микроРНК хозяина. E6 и E7 HPV-белки модулируют экспрессию ДНК-метилтрансфераз, ферментов, регулирующих экспрессию генов путем метилирования их промоторных областей [17; 18].
Целью данного обзора является анализ литературы, посвященной профилированию miRNA при переходе от предраковых заболеваний к ИРШМ.
Проведен анализ всех доступных исследований, посвященных нарушению регуляции экспрессии miRNA во время развития ИРШМ, особенно при интраэпителиальных поражениях. В период с января 2010 года по декабрь 2019 года в базе данных PubMed был осуществлен поиск публикаций с ключевыми словами «miRNA / microRNA», «рак шейки матки/ cervical cancer», «прогрессия», «CIN», «предраковые поражения». 24 исследования были отобраны и включены в настоящий обзор.
Для miRNAs, которые в двух или более исследованиях изменяли свою экспрессию, проводился поиск целевых генов, используя базу данных miRWalk 2.0 [19]. Полученные списки генов-мишеней были протестированы с использованием программного обеспечения Enrichr на их представленность в биологических сигнальных путях. Enrichr - это веб-приложение, которое включает различные библиотеки генов [20]. В частности, для данного исследования были использованы KEGG и VirusMint. Релевантность обогащения каждого набора генов оценивали по p-значению, скорректированному для множественного тестирования на основе гипергеометрического распределения [21].
Основные результаты исследований по экспрессии miRNA при прогрессировании предраковых заболеваний в рак шейки матки
Обзор 24 наиболее важных исследований представлен в таблице 1. Одни из них исследовали переход от нормального эпителия до ИРШМ, а другие сосредоточились только на интраэпителиальных поражениях. Для оценки экспрессии miRNA наиболее часто использовались следующие методы: 1) количественная ПЦР в реальном времени (18 статей), 2) технология микрочипов (5 статей) (таблица 1).
Таблица 1
Основные результаты исследований miRNA при развитии рака шейки матки
|
Ссылка |
Дизайн исследования/ кол-во образцов |
Количество анализируемых микроРНК (методика) |
Повышена экспрессия микроРНК при прогрессии |
Снижена экспрессия микроРНК при прогрессии |
||||
|
Исследования биопсийного материала |
SCC> CIN2 / 3> |
SCC> CIN2 / 3 |
CIN2 / 3> CIN1 / норма |
CIN1 / норма> CIN 2 / 3 |
CIN2 / 3> SCC |
норма / CIN 1> CIN2 / 3> SCC |
||
|
Li B. et al., 2010 [22] |
Образцы (n = 140) (только ВПЧ + или -) |
1 микроРНК |
- |
- |
- |
- |
- |
Mir-34а |
|
Li Y. et al., 2010 [23] |
Образцы (n = 78): 22 CIN1, 27 CIN2 и 29 CIN3.генотип. ВПЧ |
1 микроРНК (ОТ-ПЦР) |
- |
- |
- |
- |
- |
Mir-218 |
|
Li B.H. et al., 2011 [24HYPERLINK "https://www.ncbi.nlm.nih.gov/ pmc/articles/PMC6020348/#CR32"] |
Образцы (n = 125) |
1 микроРНК (ОТ-ПЦР) |
- |
- |
- |
- |
miR-100 |
- |
|
Deftereos G. et al., 2011 [25] |
Образцы (n = 142) |
2 микроРНК (ПЦР) |
микроРНК-21 |
|||||
|
Wang X. et al., 2014 [26] |
Образцы (n = 158): |
8 микроРНК (ПЦР) |
miR-7a |
микроРНК-16 |
miR-29а |
|||
|
Leung C. et al., 2014 [27] |
Образцы (n = 37): 10 SCC, 16 CIN2 / 3 и 11 CIN 1 |
1 микроРНК |
miR-135а |
|||||
|
Gocze K. et al., 2015 [28] |
Образцы (n = 98) |
6 микроРНК |
miR-21 |
микроРНК-196 |
miR-34a |
|||
|
Shishodia G. et al., 2015 [29] |
Образцы (n = 102) |
2 микроРНК |
miR-21 |
let- 7a |
||||
|
Zheng W. et al., 2015 [30] |
Образцы (n = 140) |
1 микроРНК |
miR-31 |
|||||
|
Ma L. et al., 2015 [31] |
Образцы (n = 163): |
1 микроРНК |
miR -146а |
|||||
|
Bumrungthai S. et al., 2015 [32] |
Образцы (n = 111): 43 SCC, 22 CIN II-III, CIN I, 12 цервицитов и 20 нормальных |
1 микроРНК |
miR-21 (без значительного увеличения между CIN II-III и CIN I) |
|||||
|
Jimenez-Wences H. et al., 2016 [33] |
Образцы (n = 49) |
3 микроРНК |
miR-193b |
miR-218 |
||||
|
Wen F. et al., 2017 [34] |
Образцы (n = 607) |
1 микроРНК |
miR-15b |
|||||
|
Sun P. et al., 2017 [35] |
Образцы (n = 165) |
1 микроРНК |
микроРНК-466 |
|||||
|
Исследования цитологического материала |
||||||||
|
Tian Q. et al., 2014 [36] |
Образцы (n = 1021) |
6 микроРНК, (ПЦР) |
miR-34a |
|||||
|
Ribeiro J. et al., 2015 [37] |
Образцы (n = 114) |
2 микроРНК (ПЦР) |
miR-34a |
|||||
|
Malta M. et al., 2015 [38] |
Образцы (n = 73): |
1 микроРНК (ПЦР) |
let-7c |
|||||
|
Исследования биопсийного материала |
||||||||
|
Pereira P.M. et al., 2010 [39] |
Образцы (n = 25): 4 SCC, 5 HSIL, 9 LSIL и 19 нормальных тканей (только HPV + или -) |
281 микроРНК (микрочипы) |
miR-10a |
miR-16 |
- |
- |
miR-522-5p |
miR-26a |
|
Li Y. et al., 2011 [40] |
Образцы (n = 18): 6 HPV-позитивных SCCs, 6 HPV -позитивных CIN2 / 3 и 6 нормальных тканей. Образцы (n = 91): 24 HPV позитивных SCCs, 24 ВПЧ-положительные CIN2 / 3 и 43 нормальные ткани, генотип. ВПЧ |
875 микроРНК (микрочип) + Проверка: 6 микроРНК |
miR-15b |
let-7b |
||||
|
Zeng K. et al., 2015 [41] |
Образцы (n = 12) Образцы (n = 103). Генотипирование ВПЧ |
866 человеческих и 89 вирусных микроРНК (микрочип) Валидация 9 микроРНК (ПЦР) |
miR-9 |
miR-195 |
||||
|
Wilting S.M. et al., 2013 [14] |
Образцы (n = 47): 10 SCC, 9 AdCAs, 18 CIN2 / 3 и 10 нормальных тканей |
472 микроРНК (микрочипы Agilent) |
let-7i |
miR-7d |
let-7 g |
miR-193a-3p |
miR-100 |
miR-134 |
|
Cheung T.H. et al., 2012 [42] |
Образцы (n = 33): образцы биопсии пациентов 12 CIN2, 12 CIN3, 9 CC и 9 нормальными клетками |
202 микроРНК (ПЦР) |
miR-9 |
miR-10a |
miR-193b |
|||
|
Bierkens M. et al., 2013 [43] |
Образцы (n = 28) |
1 миРНК |
miR-375 |
|||||
|
Villegas-Ruiz V. et al., 2014 [44] |
Образцы (n = 8) |
Обнаружение: 7788 микроРНК Валидация: 1 микроРНК (ПЦР) |
miR-196a |
|||||
Сокращения: AdCA - аденоматозная карцинома, CIS - карцинома in situ, ICC - инвазивный рак шейки матки, SCC - плоскоклеточный рак шейки матки, HSIL - плоскоклеточное интраэпителиальное поражение, FFPE - фиксированный формалином, заключенный в парафин.
Только 1 исследование качественно проанализировало экспрессию miRNA с помощью ПЦР с обратной транскрипцией (ОТ-ПЦР) [43]. Исследования, выполненные на микрочипах, различались по количеству исследованных микроРНК (от 202 до 875). В исследованиях микроРНК-кандидатов авторы анализировали только одну или несколько микроРНК на основании литературных данных или своих собственных предыдущих исследований. В нескольких исследованиях был проведен скрининг с последующей проверкой с использованием ПЦР [40; 41].
В целом между исследованиями наблюдается большая вариабельность как в отношении количества исследуемых образцов (от 12 до 1021), так и типа образцов. В большинстве исследований были проанализированы фиксированные формалином, залитые парафином ткани (FFPE) или замороженные ткани шейки матки [37; 38]. В нескольких исследованиях приведены данные о генотипировании ВПЧ или по крайней мере об определении ВПЧ-негативных и позитивных.
Первое исследование на микрочипах было опубликовано Pereira и его коллегами в 2010 году [39]: выявлено 281 микроРНК в 25 биологических образцах и показана высокая вариабельность экспрессии miRNA, особенно среди нормальных образцов. Была обнаружена 21 микроРНК со статистически значимой дифференциальной экспрессией между пулом нормальных образцов, группой CIN1 и CIN3 и образцами плоскоклеточного рака шейки матки (ПРШМ). Экспрессия 8 таких miRNAs (miR-26a, miR-29a, miR-143, miR-145, miR-99a, miR-199a, miR-203 и miR-513) постепенно снижалась, а экспрессия 5 miRNAs (miR-10a, miR-132, miR-148a, miR-196a и miR-302b) увеличивалась в этих группах. Также наблюдалось уменьшение экспрессии от нормальной ткани к CIN с последующим увеличением от CIN к SCC для 6 микроРНК (miR-16, miR-27a, miR-106a, miR-142-5p, miR-197 и miR-205) и увеличение с последующим уменьшением в 2 раза для miR-522(miR-522-5p) и miR-512-3p.
Следующее исследование на микрочипах было выполнено Li Y. и коллегами на 18 образцах, включая HPV16-позитивные ПРШМ и CIN [40]. Из 875 протестированных микроРНК 31 (14 с пониженной и 17 с повышенной) показала значительные тенденции изменения экспрессии от нормального эпителия к опухоли. 6 из них (miR-29a, miR-92a, miR-99a, miR-155, miR-195 и miR-375) были подтверждены с помощью ПЦР на 91 образце биопсии (24 ПРШМ и 24 CIN2/3, 43 образца нормальной ткани). Обнаруженное снижение экспрессии miR-99a и miR-29a подтвердило результаты Pereira et al. [39]. Экспрессия miR-218 была наиболее значительно снижена, что подтверждается и последующими исследованиями Wilting et al. [14] и Zeng et al. [41]. В исследовании, проведенном Li Y. и коллегами [23], эта miRNA также показала более низкие уровни экспрессии у пациентов с CIN2-3, чем у пациентов с CIN1. Эта микроРНК имеет сотни генов-мишеней, включая LAMB3, который увеличивает клеточную миграцию и способствует канцерогенезу на мышиной модели и в кератиноцитах человека [23]. Li Y. и коллеги [40] также сравнивали экспрессию miRNA в CIN2/3 и ПРШМ с инфекцией HPV16 с нормальными тканями шейки матки без HPV. Две группы показали значительную разницу только для miR-375 и miR-99a.
В другом исследовании Li Y. и коллег [40] уровни miR-100 были значительно ниже в CIN3, чем в CIN1 и CIN2 [14; 24]. Этот результат также был подтвержден in vitro Li B.H. et al. на клеточных линиях шейки матки [24]. Подавление miR-100 его специфическим ингибитором заметно стимулировало рост клеток, уменьшало апоптоз клеток и ускоряло прогрессирование фазы G2/M в клетках HaCaT, которые конститутивно экспрессируют высокий уровень этой miRNA. Эти результаты указывают на то, что сниженная экспрессия miR-100 может способствовать канцерогенезу шейки матки, регулируя рост клеток, клеточный цикл и апоптоз.
В исследовании Cheung и коллег экспрессию панели из 202 микроРНК исследовали в образцах нормального эпителия, CIN2, CIN3 и ИРШМ с использованием платформы qPCR [42]. 12 микроРНК (10 с повышенной и 2 с пониженной экспрессией) дифференциально экспрессировались в биопсийных образцах с CIN2/3 по сравнению с нормальными эпителиальными клетками шейки матки. Эта сигнатура из 12 микроРНК могла четко отделить CIN2/3 от нормальной ткани в независимой группе образцов (6 CIN2, 18 CIN3, 9 нормальных) и пациентов с ПРШМ (51) от лиц с нормальным эпителием шейки матки, но не CIN2 от CIN3. В целом miR-20b и miR-9 показали наивысший уровень экспрессии. Экспрессия miR-9 была также значительно выше при ПРШМ, чем при CIN2/3. Авторы считают, что miR-9 представляет особый интерес, потому что она стимулирует ангиогенез в зависимости от типа клеток [42]. Было показано, что miR-9, опосредованно понижая экспрессию E-кадгерина, приводит к активации β-катенина, что в свою очередь приводит к усилению регуляции целевого гена VEGFA, проангиогенного фактора [45]. Как при CIN, так и при ПРШМ наблюдали подавление экспрессии E-cadherin, что согласуется с описанным выше повышением miR-9 при CIN и ПРШМ [42].
Уилтинг и соавторы [14] исследовали с помощью микрочипов экспрессию 472 miRNAs у 47 субъектов (нормальный эпителий, CIN2/3, ПРШМ) и сравнивали их дифференциальную экспрессию с гистологией. В общей сложности 106 микроРНК были дифференциально экспрессированы при CIN2/3 и ПРШМ по сравнению с нормальным эпителием. Некоторые из этих miRNA подтвердили результаты Pereira et al. [39] и Cheung et al. [42], а также наблюдения Li et al. [14; 40] по увеличению miR-155 при переходе от CIN1 к ПРШМ. Zeng K. и его коллеги [41] сравнили профили экспрессии miRNA при ИРШМ, CIN и в нормальных тканях шейки матки с помощью микрочипового анализа и обнаружили, что экспрессия некоторых miRNAs значительно нарушена. В частности, 16 miRNAs имели повышенную экспрессию, а 10 значительно сниженную при сравнении ПРШМ с нормальной тканью. miR-21, miR-21-3p, miR-15b и miR-16 были наиболее сверхэкспрессированными, тогда как miR-218 и miR-376 были наиболее гипоэкспрессированными. 44 miRNAs были дифференциально экспрессированы (13 повышено и 31 понижено) при сравнении CIN2/3 с нормальной тканью шейки матки. При сравнении CIN1 с нормальной тканью дифференциально экспрессированных микроРНК обнаружено не было. 9 miRNAs (miR-21, miR-218, miR-376a, miR-31, miR-630, miR-9, miR-195, miR-497, и miR-199b-5p), дифференциально экспрессированные в образцах шейки матки, были подтверждены с помощью ПЦР в 103 образцах с аналогичными характеристиками. Экспрессия miR-21, miR-31 и miR-9 была значительно повышена при ИРШМ, как обнаружено и в других работах [25-27; 40]. Методом ПЦР также было подтверждено, что экспрессия miR-218, miR-195, miR-497 и miR-199b-5p значительно снижена при ИРШМ и CIN2/3, а miR-376a только при ИРШМ, но не в CIN2 / 3. Однако при этом не наблюдалось дифференциальной экспрессии miRNA между CIN1 и нормальной тканью. Авторы выдвинули гипотезу, что miRNAs с пониженной экспрессией как при ПРШМ, так и при CIN2/3 могут быть вовлечены в аномальное превращение предраковых поражений в рак.
В других исследованиях изучали микроРНК, ранее идентифицированные как незначимые в развитии ИРШМ. Villegas-Ruiz и соавторы [46] после исследования профилей miRNA в опухолях по сравнению со нормальной тканью с помощью микрочипов сосредоточили внимание на экспрессии miR-196a в отношении её роли в прогрессировании ИРШМ: показали увеличение экспрессии этой miRNA при переходе от нормальных тканей к CIN и опухолям, подтверждая выводы Pereira et al. В исследовании Gocze [28] экспрессия miR-196a также увеличивалась при CIN1 и CIN2/3, но была значительно ниже при ПРШМ, чем при CIN2/3.
Leung с коллегами обнаружили сверхэкспрессию miR-135a при ПРШМ по сравнению с CIN1 и CIN2/3 в образцах FFPE блоков [27]. Авторы показали, что miR-135a регулирует передачу сигналов Wnt/β-catenin через гены-мишени SIAH1 и, возможно, APC . Это может в конечном итоге вызвать трансформацию в ИРШМ, при этом miR-135a также регулирует миграцию и пролиферацию раковых клеток.
Li B. et al. [22] исследовали экспрессию pri-miR-34a, предшественника miR-34a, в нормальном эпителии шейки матки, CIN и ИРШМ с помощью полуколичественной ОТ-ПЦР. Экспрессия pri-miR-34a была значительно снижена в группах CIN и ИРШМ по сравнению с нормальным эпителием. Снижение экспрессии pri-miR-34a, связанное с инфекцией HPV, происходило до морфологических нарушений эпителия шейки матки и до изменений, индуцированных HR-HPV E6 в р53-зависимом сигнальном пути. Таким образом, авторы пришли к выводу, что это начальное событие в развитии ИРШМ, и что miR-34a и его предшественник могут рассматриваться как потенциальные молекулярные маркеры для скрининга и мишени для блокирования развития ИРШМ.
Дифференциально экспрессирующиеся микроРНК и их гены-мишени
Список микроРНК, для которых установлено повышение экспрессии (17) или понижение экспрессии (13) при прогрессировании ИРШМ в нескольких исследованиях, приведен в таблице 2. Среди сверхэкспрессированных микроРНК: 1) по данным 5 исследований - одна (miR-21), 2) по данным 3 других исследований - 9 (miR-9, miR-16, miR-25, miR-10a, miR-20b, miR-31 miR- 92a, miR-106a и miR-155) и 3) по данным 2 исследований - 8 (miR-15b, miR-17, miR-27a, miR-92b, miR-93, miR-146a, miR-185 и miR-196a) связаны с канцерогенезом шейки матки. Среди гипоэкспрессированных miRNAs подобная ассоциация была обнаружена для miR-218 в 6 исследованиях, для miR-375 и miR-203 в 4 исследованиях и для miR-99a, miR-29a, miR-195, miR-125b, miR-34a и miR-100 в 3 исследованиях. На рисунке 1 представлена информация о совпадении данных разных исследований.
Таблица 2
miRNAs, участвующие в развитии РШМ, выявленные в более чем одном исследовании
|
микроРНК |
Ссылка на исследование |
микроРНК |
Ссылка на исследование |
|
miR-15b a , miR-17, miR-92b, miR-93, miR-185, miR-16, miR-106a |
[14; 40] |
miR-218 |
[14; 23; 36; 40; 41] |
|
miR-92a, miR-25 |
[14; 26] |
miR-375 |
[14; 36; 40; 43] |
|
miR-21 |
[25; 29; 32; 41] |
miR-99а |
[14; 39; 40] |
|
miR-10a |
[14; 39; 42] |
miR-29а |
[26; 39; 40] |
|
miR-20b |
[14; 40; 42] |
miR-203 |
[14; 28; 39; 42] |
|
miR-155 |
[14; 38; 40] |
miR-195 |
[14; 40; 41] |
|
miR-9 |
[14; 41; 42] |
miR-125b |
[14; 37; 40] |
|
miR-31 |
[30; 40; 41] |
miR-34а |
[28; 36; 37] |
|
miR-27а |
[28; 39] |
miR-100 |
[14; 24; 40] |
|
miR-146а |
[14; 31] |
miR-145 |
[39; 40] |
|
miR-196a |
[39; 44] |
miR-424 |
[36; 40] |
|
miR-193b b |
[14; 42] |
miR-497 |
[14; 41] |
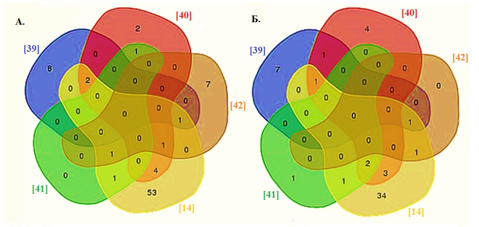
Рис. 1. Диаграмма Венна, показывающая (A) повышенную и (Б) пониженную экспрессию miRNAs в разных исследованиях
Количество экспериментально подтвержденных генов-мишеней для вышеупомянутых микроРНК, полученных в базе данных miRWalk 2.0, представлено в таблице 3.
Около 16671 генов-мишеней было идентифицировано для гиперэкспрессированных микроРНК и 7075 генов для микроРНК с пониженной экспрессией. После удаления из этого перечня генов с множественными сайтами связывания miRNA, а также генов, общих между гиперэкспрессированными микроРНК и микроРНК с пониженной экспрессией, осталось 3395 и 1575 генов соответственно. Анализ обогащения генов-мишеней для микроРНК с пониженной экспрессией обеспечил статистически значимые данные об избыточной репрезентации и Kegg биологических путях (таблица 4). Впоследствии анализ обогащения был сосредоточен только на генах-мишенях микроРНК, обнаруженных в большом количестве исследований (элементы Kegg pathway и Virus Mint).
Таблица 3
Гены-мишени для miRNAs, участвующих в прогрессии ИРШМ (данные из miRWalk 2.0)
|
Гены-мишени miRNA со сниженной экспрессией (n) |
Гены-мишени miRNA с повышенной экспрессией (n) |
Гены-мишени (без множественных сайтов-мишеней) miRNA со сниженной экспрессией (n) |
Гены-мишени (без множественных сайтов-мишеней) miRNA с повышенной экспрессией (n) |
Уникальные гены-мишени miRNA со сниженной экспрессией |
Уникальные гены-мишени miRNA с повышенной экспрессией |
|
7075 |
16671 |
3584 |
5987 |
1576 |
3564 |
Таблица 4
Анализ обогащения для генов-мишеней микроРНК, участвующих в прогрессии ИРШМ (Kegg pathway и Virus Mint)
|
№ |
Kegg pathway |
P-value |
Z-score |
Virus Mint |
P-value |
Z-score |
|
1 |
Viral carcinogenesis_Homo sapiens |
9.94E-2 |
-1.98 |
Epstein-Barr virus (strain GD1) |
8.24E-13 |
-1.19 |
|
2 |
Pathways in cancer_Homo sapiens |
9.51E-1 |
-2.06 |
Human immunodeficiency virus 1 |
2.13E-08 |
-0.93 |
|
3 |
Proteoglycans in cancer_Homo sapiens |
5.83E-1 |
-1.99 |
Homo sapiens |
5.09E-07 |
-0.53 |
|
4 |
Hepatitis B_Homo sapiens_hsa05161 |
1.58E-0 |
-1.86 |
Human papillomavirus type 16 |
3,19E-05 |
-0.49 |
|
5 |
HTLV-I infection_Homo sapiens_hsa05166 |
1.78E-10 |
-1.82 |
Human adenovirus 5 |
2,57E-04 |
-0.07 |
|
6 |
Cell cycle_Homo sapiens_hsa04110 |
1.74E-10 |
-1.63 |
Human papillomavirus type 11 |
7,22E-04 |
-0.03 |
|
7 |
Prostate cancer_Homo sapiens_hsa05215 |
3.60E-08 |
-1.8 |
Human immunodeficiency virus type 1 |
1,12E-03 |
1.17 |
|
8 |
Endocytosis_Homo sapiens_hsa04144 |
5.14E-08 |
-1.77 |
Human adenovirus 2 |
2,55E-03 |
1.46 |
|
9 |
Chronic myeloid leukemia_Homo sapiens |
5.13E-08 |
-1.71 |
Simian virus 40 |
6,24E-03 |
1.29 |
|
10 |
Epstein-Barr virus infection_Homo sapiens |
6.12E-08 |
-1.73 |
Vaccinia virus |
8,74E-03 |
3.98 |
Потенциальные гены-мишени miRNA были также протестированы в базе данных CCDB, где перечислены 257 генов с повышенной экспрессией и 110 со сниженной экспрессией при ИРШМ. Среди тех генов, которые ранее были идентифицированы как мишени для микроРНК с пониженной экспрессией (соответственно сами гены должны быть сверхэкспрессированы при прогрессировании до ИРШМ), 22 (8,5%) действительно были сверхэкспрессированы при ИРШМ. С другой стороны, среди генов-мишеней микроРНК с повышенной экспрессией (соответственно эти гены должны быть слабо экспрессированы при развитии ИРШМ) 34 (30,9%) действительно были гипоэкспрессированы при ИРШМ (таблица 5).
Таблица 5
Гены-мишени miRNAs при развитии ИРШМ
|
Гиперэкспрессированные гены (n=22) |
Гипоэкспрессированные гены (n=34) |
||||
|
APLP2 |
KLK2 |
ANLN |
ITM2C |
NDUFS6 |
VEGFC |
|
CTSS |
KRT10 |
APOL2 |
KIT |
PCNA |
|
|
DAP3 |
MEF2A |
BCL2L1 |
KRT7 |
PDGFRA |
|
|
EPB41L4B |
NTN4 |
CEACAM6 |
KRT8 |
PIGF |
|
|
GARS |
OAT |
CRABP2 |
MAP3K11 |
PIGT |
|
|
GATA3 |
PA2G4 |
CTGF |
MCM2 |
S100A8 |
|
|
HYAL1 |
PDIA3 |
ERBB4 |
MMP1 |
SAA1 |
|
|
KLF3 |
RTN3 |
FGFR2 |
MMP12 |
SFN |
|
|
THBS1 |
SERPINB5 |
IER5 |
MMP14 |
STAT1 |
|
|
TIMP3 |
SMAD2 |
IFI6 |
MYCN |
TBC1D1 |
|
|
TGFB1 |
IMP3 |
NDRG1 |
TPX2 |
||
Таким образом, в разных исследованиях неоднократно было обнаружено, что уровни экспрессии нескольких miRNAs связаны с развитием предраковых поражений в ИРШМ (рисунок 2).
Гиперэкспрессированная микроРНК miR-21, ассоциированная с CIN и ИРШМ в 4 исследованиях [14; 25; 41], представляет особый интерес. miR-21 является одной из наиболее широко распространенных микроРНК у млекопитающих. Её активация связана со многими видами рака, различного происхождения [47]. Определение функции miR-21 было объектом многих исследовательских групп за последние 10 лет. miR-21 взаимодействует с большим количеством генов-мишеней и тонко реагирует на внеклеточные сигналы. Хотя miR-21 был признан ключевым регулятором многих процессов, вовлеченных в контроль пролиферации клеток, он также связан с ключевыми процессами при воспалении. В отличие от других микроРНК, miR-21 не просто характерен для провоспалительного или иммуносупрессивного статуса, но он действует как ключевой сигнал, опосредующий их баланс [47]. Другие микроРНК, изменение экспрессии которых показано в данном обзоре (miR-125b, miR-155, miR-146a), играют важную роль в иммунном ответе и воспалении [48]. Например, при развитии ИРШМ наблюдается пониженная экспрессия miR-125b. Интересно, что эта miRNA играет важную роль в иммунном ответе и воспалении, но может действовать как онкоген и онкосупрессор. miR-125b связан с HPV-индуцированным канцерогенезом по двум сигнальным путям: a) имеет гомологию с HPV-L2, которая необходима для сборки вирусного капсида (поэтому после ранней инфекции miR-125b может ингибировать репликацию HPV); б) miR-125b приводит к инактивации сигнального пути p53, таким образом, поддерживая клетки, пораженные вирусом, жизнеспособными. Это может привести к повышенному риску интеграции ВПЧ-генома и развитию опухолевого поражения [16].
Обнаружено, что miR-34a, miR-125 и miR-375 снижают экспрессию при переходе от нормального эпителия к ИРШМ как в тканях, так и в клетках цитологических мазков. Это открывает возможность применения этих микроРНК в процессе тестирования больших групп населения. Основным ограничением существующих исследований является ограниченное число включенных в них объектов. В принципе, только образцы, включающие все поражения, выявленные при популяционном скрининге, и случайную выборку здоровых женщин из той же группы, должны отбираться для подобных исследований. Вместо этого большинство исследований проводились на вспомогательных материалах, происхождение и принцип выбора которых подробно не описаны. При этом экспрессия одних микроРНК была дизрегулирована на всех стадиях канцерогенеза, других только при переходе от CIN2/3 к ИРШМ [14; 42] (рисунок 2). При изучении роли экспериментально подтвержденных генов-мишеней микроРНК, идентифицированных как изменяющие экспрессию при канцерогенезе шейки матки, было обнаружено много сигнальных путей, имеющих отношение к возникновению и прогрессированию ИРШМ. Интересно, что совпадение данных исследований на основе микрочипов было ограниченным. Это может быть связано с различиями в дизайне исследования и группах пациентов (разные этнические группы) или с небольшим количеством исследованных образцов ИРШМ и CIN. Также во всех исследованиях использовались разные микрочипы, содержащие различное количество и типы зондов miRNA. Из наиболее важных микроРНК с нарушением экспрессии, определённых в многочисленных исследованиях, следует отметить miR-10a [14; 42], miR-20b [14; 40], miR-9 [14; 41; 42], miR-16 и miR-106a [14; 39] (с повышенной экспрессией) и miR-99a [14; 39; 40], miR-203 [14; 39; 42] и miR-195 [14; 40; 41] (с пониженной экспрессией). Несмотря на быстро растущее число обнаруженных микроРНК (в настоящее время более 2600), большинство исследований сосредоточено на первых 800 микроРНК. В последнее время возросло использование технологии секвенирования следующего поколения (NGS) для профилирования экспрессии miRNA. Секвенирование малых РНК, позволяющее также обнаруживать другие небольшие некодирующие РНК (пиРНК, тРНК), обладает наибольшей чувствительностью обнаружения и высочайшей точностью в анализе дифференциальной экспрессии. Для применения методов NGS к малым РНК стандартизация экспериментальных и биоинформационных протоколов только началась. Тем не менее в ближайшем будущем ожидается значительное увеличение числа исследований, посвященных применению NGS к miRNAs, в том числе при канцерогенезе шейки матки. Идеальным подходом было бы применение NGS в однородных крупномасштабных исследованиях для исследования всего «miRNome» (всей совокупности микроРНК, по аналогии с геномом) с целью выявления надежных miRNA-кандидатов, релевантных для прогрессирования ИРШМ [49].
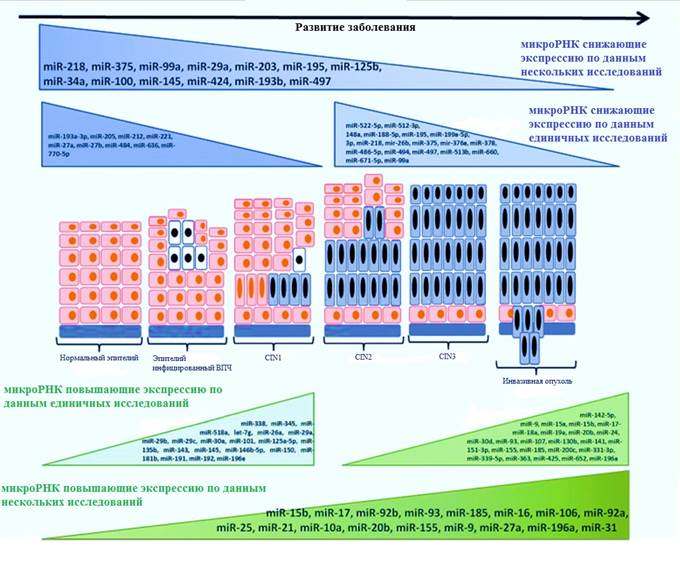
Рис. 2. Сводная информация о нарушении экспрессии miRNAs при развитии рака шейки матки
Заключение. Важная проблема скрининга ИРШМ заключается в том, что пока нет однозначного ответа на вопрос, могут ли определенные сигнатуры miRNA быть ассоциированы с риском CIN высокой степени. Исследование уровней miRNA в клетках шейки матки с цитологических препаратов, безусловно, открывает новые перспективы применения молекулярных маркеров в программах скрининга.
Библиографическая ссылка
Димитриади Т.А., Бурцев Д.В., Дженкова Е.А., Кутилин Д.С. МИКРОРНК КАК МАРКЕРЫ ПРОГРЕССИРОВАНИЯ ПРЕДРАКОВЫХ ЗАБОЛЕВАНИЙ В РАК ШЕЙКИ МАТКИ // Современные проблемы науки и образования. 2020. № 1. ;URL: https://science-education.ru/ru/article/view?id=29529 (дата обращения: 31.01.2026).
DOI: https://doi.org/10.17513/spno.29529



